分类特征的 SHAP
将经过One-Hot 编码转换的分类特征的 SHAP 值相加
可直接在橱窗里购买,或者到文末领取优惠后购买:
分类特征需要先进行转换,然后才能用于模型。One-Hot 编码是一种常见的方法:我们最终会得到每个类别的二进制变量。这很好,直到理解使用 SHAP 的模型为止。每个二进制变量都有自己的 SHAP 值。这使得很难理解原始分类特征的整体贡献。
一种简单的方法是将每个二进制变量的 SHAP 值加在一起。这可以解释为原始分类特征的 SHAP 值。我们将向你介绍执行此操作的 Python 代码。我们将看到我们能够使用 SHAP 聚合图。但是,在理解分类特征关系的性质时,这些是有限的。所以,最后我们向你展示如何使用箱线图来可视化 SHAP 值。
如果你不熟悉 SHAP 或 Python 包,我建议你阅读一下之前的这篇文章《[[Python 中的 SHAP 简介]]》。我们将深入探讨如何解释 SHAP 值。我们还探讨了本文中使用的一些聚合。
处理分类变量时还有另一种解决方案。那就是使用 CatBoost 进行建模。关于 CatBoost 如何进行建模,我以后会写一篇相关文章《[使用 CatBoost 实现分类特征的 SHAP]》,你可以在其中找到此解决方案。
数据集
为了演示分类特征的问题,我们将使用蘑菇分类数据集。你可以在图 1 中看到此数据集的快照。目标变量是蘑菇的类别。也就是说,蘑菇是有毒 (p) 还是可食用 (e)。你可以在UCI 的 MLR1中找到此数据集。
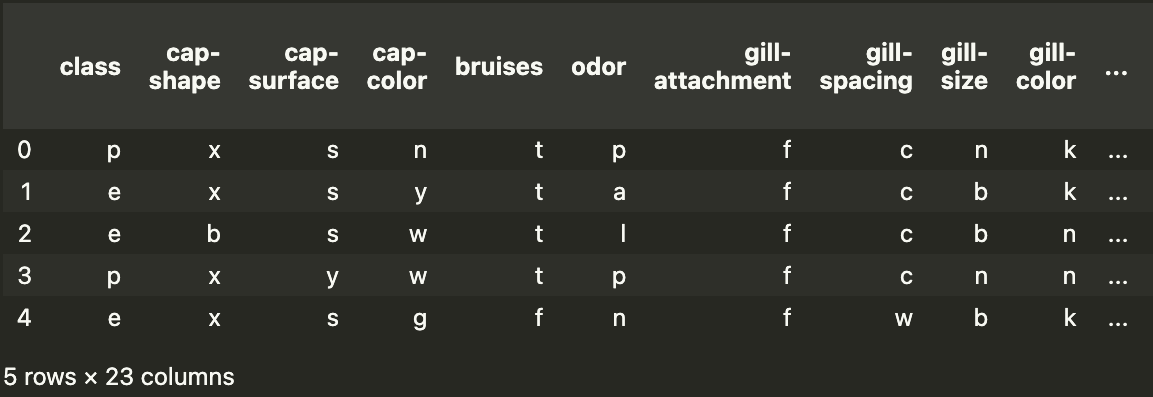
对于模型特征,我们有 22 个分类特征。对于每个特征,类别都用一个字母表示。例如,气味有 9 个独特的类别 - almond (a)、anise (l)、creosote (c), fishy (y), foul (f), musty (m), none (n), pungent (p), spicy (s)。这就是蘑菇的气味。
建模
我们将向你介绍用于分析此数据集的代码,你可以在GitHub2上找到完整的脚本。首先,我们将使用下面的 Python 包。我们有一些用于处理和可视化数据的常用包(第 1-5 行)。我们使用 OneHotEncoder 来转换分类特征(第 7 行)。我们使用 xgboost 进行建模(第 10 行)。最后,我们使用 shap 来了解我们的模型是如何工作的(第 12 行)。
1 | |
我们导入数据集(第 2 行)。我们需要一个数值目标变量,因此我们通过设置
toxicous = 1 和 edible = 0 来转换它(第 6 行)。我们还获得了分类特征(第
7 行)。我们不使用 X_cat 进行建模,但它以后会派上用场。
1 | |
要使用分类特征,我们还需要对其进行转换。我们首先安装一个编码器(第 2-3 行)。然后我们使用它来转换分类特征(第 6 行)。对于每个分类特征,每个类别都有一个二进制特征。我们为每个二进制特征创建特征名称(第 9 至 10 行)。最后,我们将它们放在一起以创建特征矩阵(第 12 行)。
1 | |
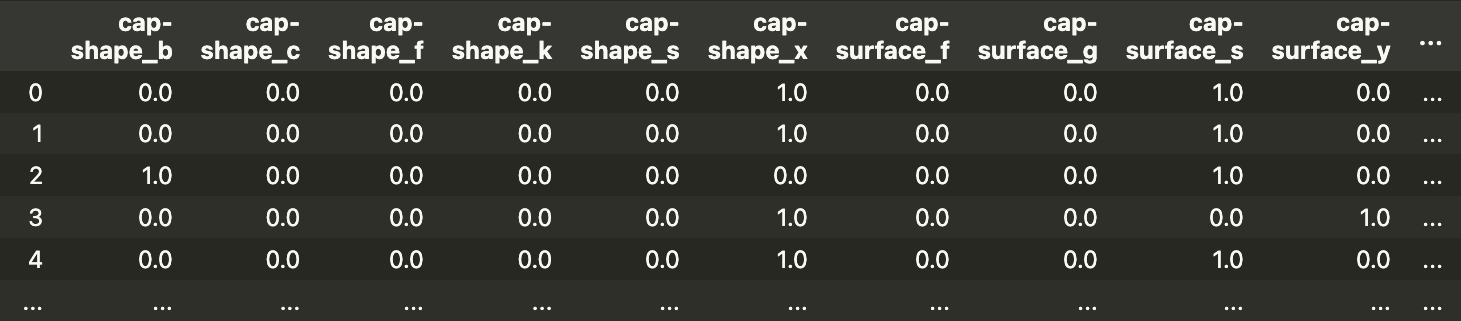
最终,我们得到了 117 个特征。你可以在图 2
中看到特征矩阵的快照。例如,你可以看到 cap-shape 现已转换为
6 个二进制变量。特征名称末尾的字母来自原始特征的类别。
我们使用此特征矩阵训练模型(第 2-5 行)。我们使用 XGBClassifier。XGBoost 模型由 10 棵树组成,每棵树的最大深度为 2。该模型在训练集上的准确率为 97.7%。
1 | |
标准 SHAP 值
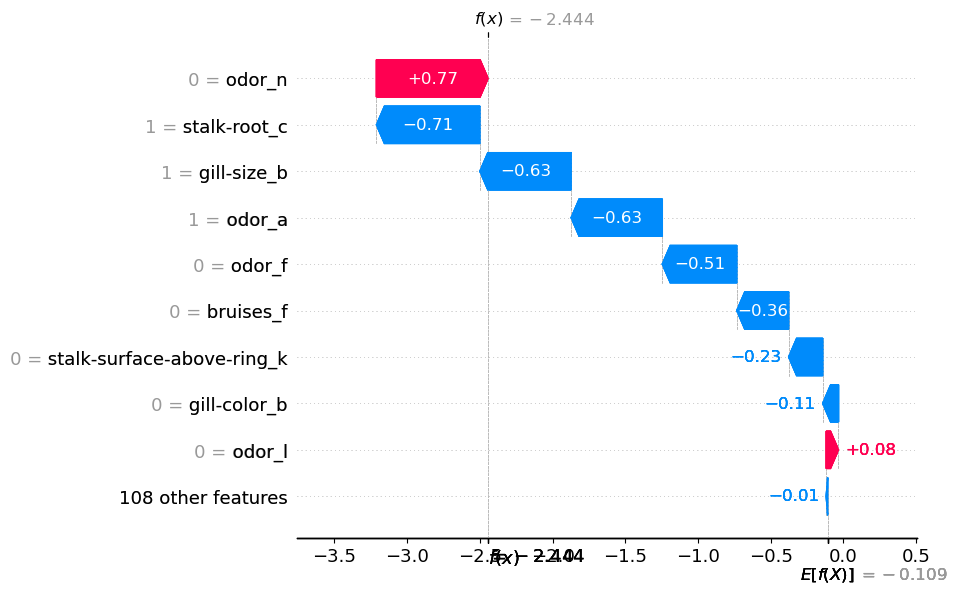
此时,我们想了解模型是如何做出这些预测的。我们首先计算 SHAP 值(第 2-3 行)。然后,我们使用瀑布图(第 6 行)可视化第一个预测的 SHAP 值。你可以在图 3 中看到此图。
1 | |
你可以看到每个二元特征都有自己的 SHAP 值。以气味为例。它在瀑布图中出现了 4 次。odor_n = 0 增加了蘑菇有毒的概率。同时,odor_a = 1、odor_f = 0 和 odor_I = 0 都降低了概率。蘑菇气味的总体贡献是什么尚不清楚。在下一节中,我们将看到,当我们将所有单独的贡献加在一起时,它确实变得清晰起来。
分类特征的 SHAP
让我们从探索shap_values对象开始。我们在下面的代码中打印该对象。你可以在下面的输出中看到它由
3 个组件组成。我们有每个预测的SHAP 值 ( values
)。数据给出了二进制特征的值。每个预测也将具有相同的基值 (
base_values )。这是平均预测对数概率。
1 | |
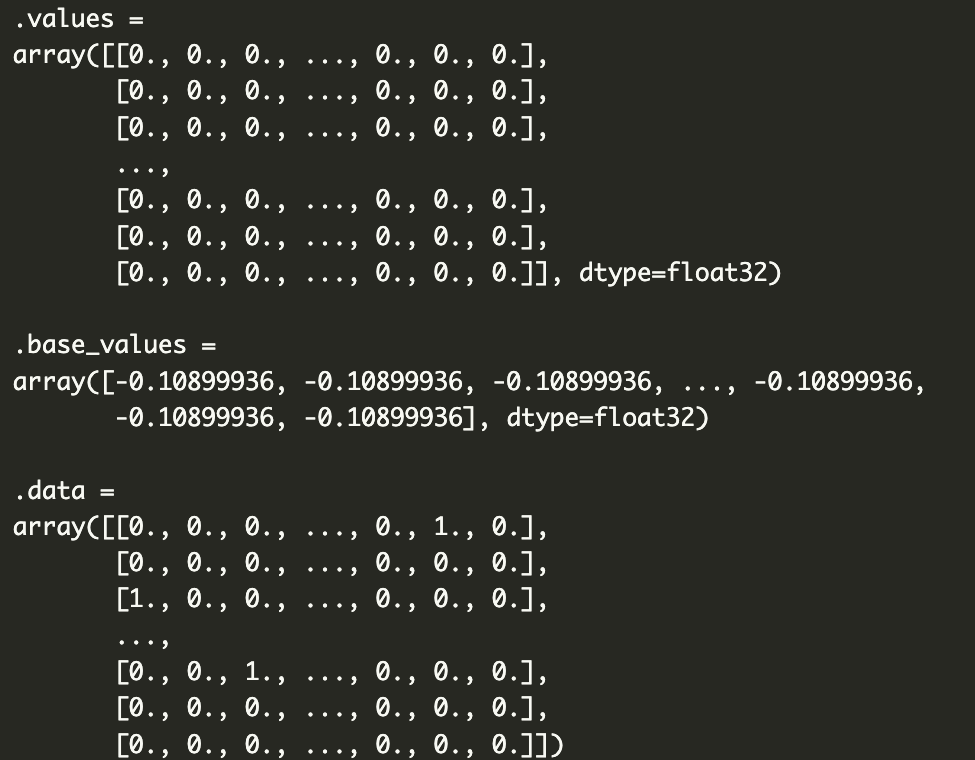
我们可以通过在下面打印它们来仔细查看第一个预测的 SHAP 值。一共有 117 个值。每个二进制变量一个。SHAP 值的顺序与 X 特征矩阵相同。请记住,第一个分类特征cap-shape有 6 个类别。这意味着前 6 个 SHAP 值对应于此特征的二进制特征。接下来的 4 个对应于cap-surface特征,依此类推。
1 | |
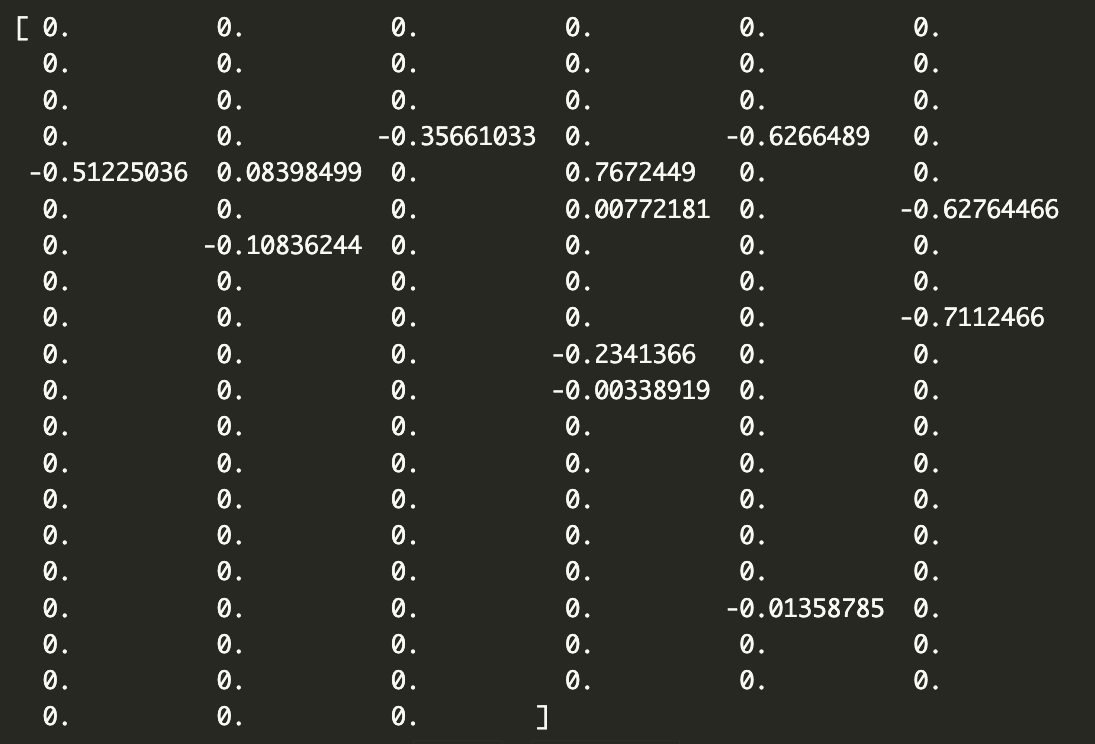
我们希望将每个分类特征的 SHAP
值加在一起。为此,我们首先创建n_categories数组。它包含每个分类变量的唯一类别数。数组中的第一个数字将是
6(表示帽形),然后是 4(表示帽表面),依此类推……
1 | |
我们使用n_categories来拆分 SHAP 值数组(第 5
行)。我们最终得到一个子列表。然后我们对每个子列表中的值求和(第 8
行)。通过这样做,我们将 SHAP 值从 117 个减少到 22
个。我们对shap_values对象中的每个观察值都执行此操作(第 2
行)。对于每次迭代,我们将求和的 shap
值添加到new_shap_values数组(第 10 行)。
1 | |
现在,我们需要做的就是用新值替换原始 SHAP 值(第 2
行)。我们还用原始分类特征中的类别字母替换二进制特征数据(第 5-6
行)。最后,我们用原始特征名称替换二进制特征名称(第 9
行)。重要的是将这些新值分别作为数组和列表传递。这些是
shap_values 对象使用的数据类型。
1 | |
更新后的 shap_values
对象可以像原始对象一样使用。在下面的代码中,我们绘制了第一次观察的瀑布图。你会注意到此代码与之前完全相同。
1 | |
你可以在图 4 中看到输出。我们现在有 22 个 SHAP 值。你还可以看到左侧的特征值已被类别标签替换。我们之前讨论过气味特征。现在你可以清楚地看到此功能的整体贡献。它使对数概率降低了 0.29。
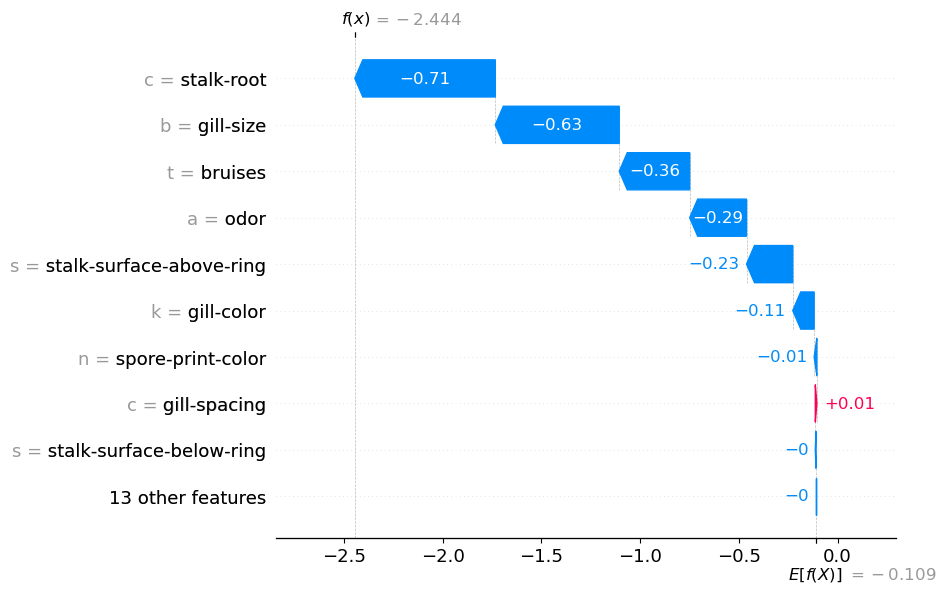
在上图中,我们得到气味 = a。这告诉我们蘑菇有“杏仁”气味。我们应避免将该图解释为“杏仁气味降低了对数几率”。我们将多个 SHAP 值相加。因此,我们应该将其解释为“杏仁气味和缺乏其他气味降低了对数几率”。例如,查看第一个瀑布图,缺乏“恶臭”气味(odor_f = 0)也降低了对数几率。
在我们继续聚合这些新的 SHAP 值之前,值得讨论一些理论。我们能够使用
SHAP
值做到这一点的原因是由于它们的可加性。也就是说,平均预测(E[f(x)])加上所有
SHAP 值等于实际预测(f(x))。通过将一些 SHAP
值加在一起,我们不会干扰此属性。这就是为什么 f(x) = -2.444
在图 3 和图 4 中相同的原因。
平均 SHAP
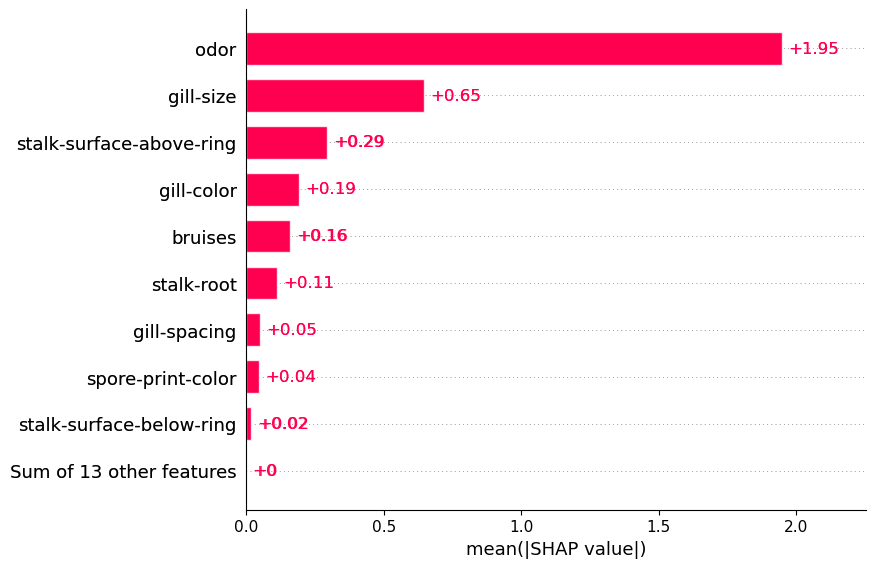
与瀑布图一样,我们可以像使用原始 SHAP 值一样使用 SHAP 聚合。例如,我们在下面的代码中使用平均 SHAP 图。查看图 5,我们可以使用此图突出显示重要的分类特征。例如,我们可以看到气味往往具有较大的正/负 SHAP 值。
1 | |
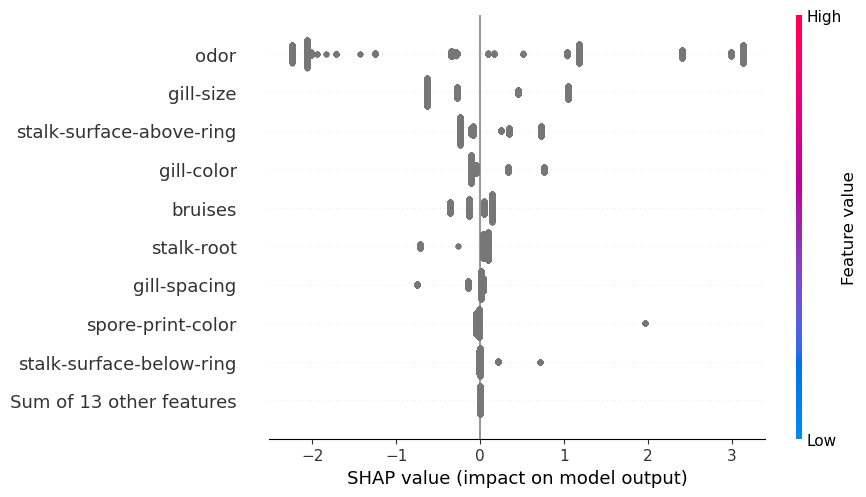
蜂群
另一种常见的聚合是蜂群图。对于连续变量,此图很有用,因为它可以帮助解释关系的性质。我们可以看到 SHAP 值如何与特征值相关联。但是,对于分类特征,我们用标签替换了特征值。因此,在图 6 中,你可以看到 SHAP 值都被赋予了相同的颜色。我们需要创建自己的图来了解这些关系的性质。
1 | |
SHAP 箱线图
我们可以采用一种方式来做到这一点,即使用 SHAP 值的箱线图。在图 7 中,你可以看到一个气味特征的箱线图。在这里,我们根据气味类别对气味特征的 SHAP 值进行了分组。你可以看到,难闻的气味会导致更高的 SHAP 值。这些蘑菇更有可能有毒。请不要吃任何有异味的蘑菇!同样,没有气味的蘑菇更有可能可以食用。一条橙色线表示这些蘑菇的所有 SHAP 值都相同。
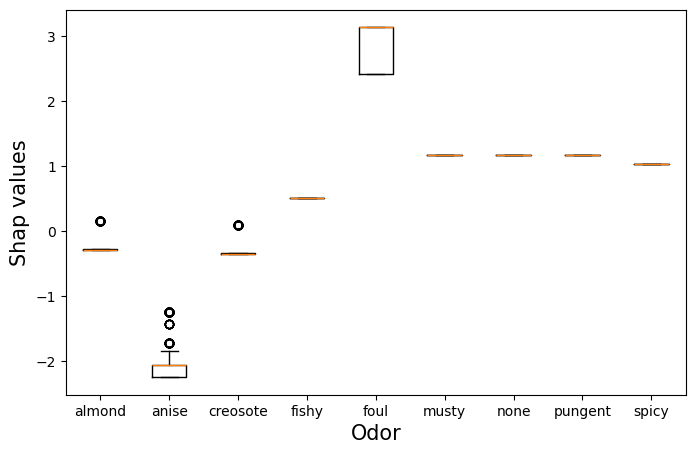
我们使用以下代码创建此箱线图。我们首先获取气味 SHAP 值(第 2 行)。请记住,这些是更新值。对于每个预测,气味特征只有一个 SHAP 值。我们还获取气味类别标签(第 3 行)。我们根据这些标签分割 SHAP 值(第 6-12 行)。我们使用这些值为每个气味类别绘制一个箱线图(第 28-33 行)。为了使图表更易于解释,我们还用完整的类别名称替换了字母(第 15-25 行)。
1 | |
实际上,你的特征中可能只有少数是分类的。你需要更新上述过程以仅对分类特征求和。你还可以想出自己的方法来可视化这些特征之间的关系。如果你想出了另一种方法,我很乐意在评论中听到它。
我还想了解特征依赖关系将如何影响此分析。根据定义,转换后的二进制特征将是相关的。这会影响 SHAP 值的计算。我们正在使用 TreeSHAP 来估计 SHAP 值。我的理解是,这些值不像 KernelSHAP 那样受依赖关系的影响。我很想在评论中听听你的想法。
参考
S. Lundberg,SHAP Python 包(2021) , https://github.com/slundberg/shap
S. Lundberg 和 S. Lee,《解释模型预测的统一方法》 (2017),https://arxiv.org/pdf/1705.07874.pdf
希望这篇文章对你有所帮助!你还可以阅读我的其他文章,或者查看有关企业 AI 实战项目的教程,相信会让你拥有更多收获。
「AI秘籍」系列课程:

分类特征的 SHAP
